Primeiro veio o Molnupiravir-agora há Paxlovid como um inibidor de protease SARS-COV-2. Uma análise NCI do ligante.
No início deste ano, Molnupiravir Chegue às manchetes como uma droga antiviral promissora. Isso agora é seguido por Paxlovidque é a primeira molécula pequena a ser apontada pelo projeto na proteína SAR-CoV-2 e que é relatada como reduzindo muito o risco de hospitalização ou morte quando administrado dentro de três dias após os sintomas que aparecem em pacientes de alto risco.
A página da Wikipedia (criada pela primeira vez em 2021) Exibirá um bom modelo JSMOL 3D deste; As coordenadas sendo geradas automaticamente em tempo de mosca a partir de uma corda de sorrisos, que especifica apenas quais átomos estão conectados na estrutura por ligações. Dado que a estrutura desta molécula incorporada na protease principal SARS-CoV-2 (citação) 10.1007/s13238-021-00883-2 (/cite) foi determinada (e pode ser vista aqui), Pensei em exibir essas coordenadas como uma alternativa à estrutura gerada pela Wikipedia/JSMOL.
Clique para obter o modelo 3D
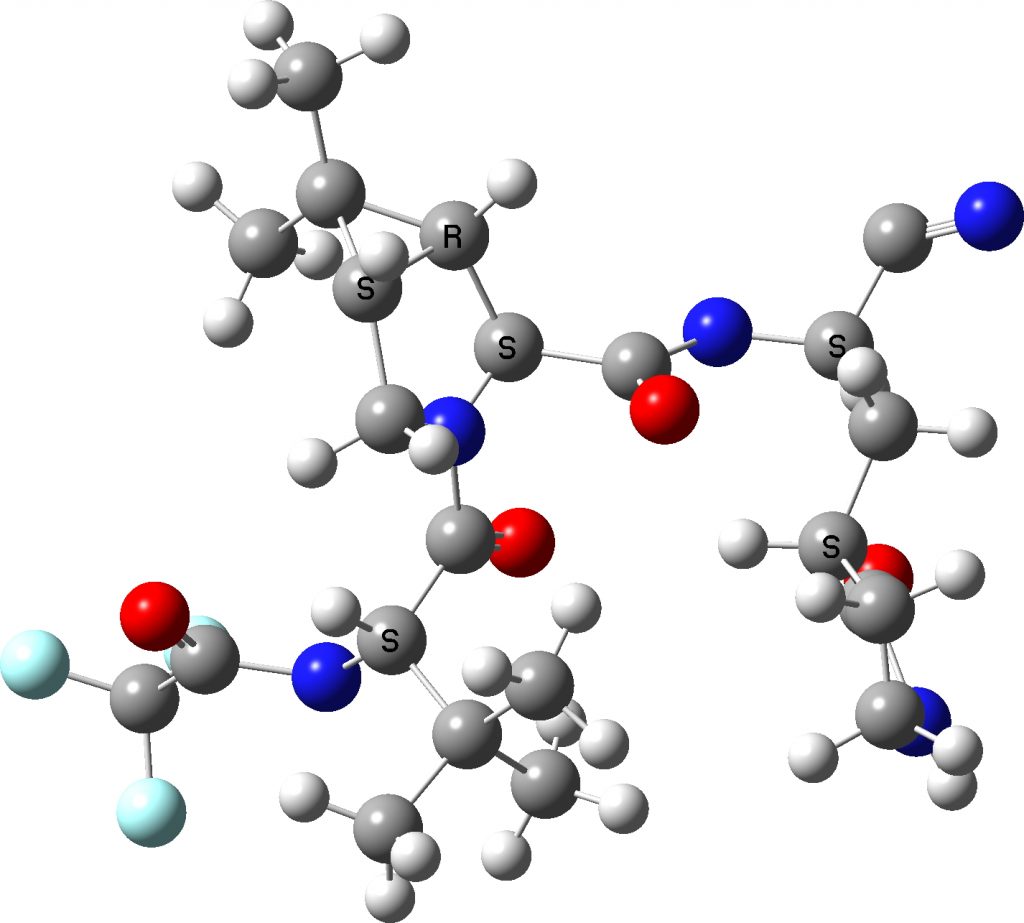
Extraí o ligante do arquivo PDF e adicionei hidrogênios manualmente para obter o resultado acima.‡ Existem dois pontos notáveis sobre essas representações:
- Um mistério diz respeito ao grupo nominal C≡N no canto superior direito, que exibe um ângulo no carbono de 117 °. Um grupo de ciano é obviamente linear (180 °). Isso não é um defeito da determinação da estrutura do cristal, mas uma indicação de uma interação bastante mais forte ocorre (como de fato observado (cite) 10.1007/S13238-021-00883-2 (/cite)). A distância entre o carbono do grupo ciano e um enxofre adjacente é de 1,814å, o que indica que uma ligação covalente se formou para o grupo ciano. O nitrogênio do grupo Cyano está 3,013å, longe de um grupo NH adjacente, o que sugere que ele é estabilizado por uma ligação de hidrogênio.
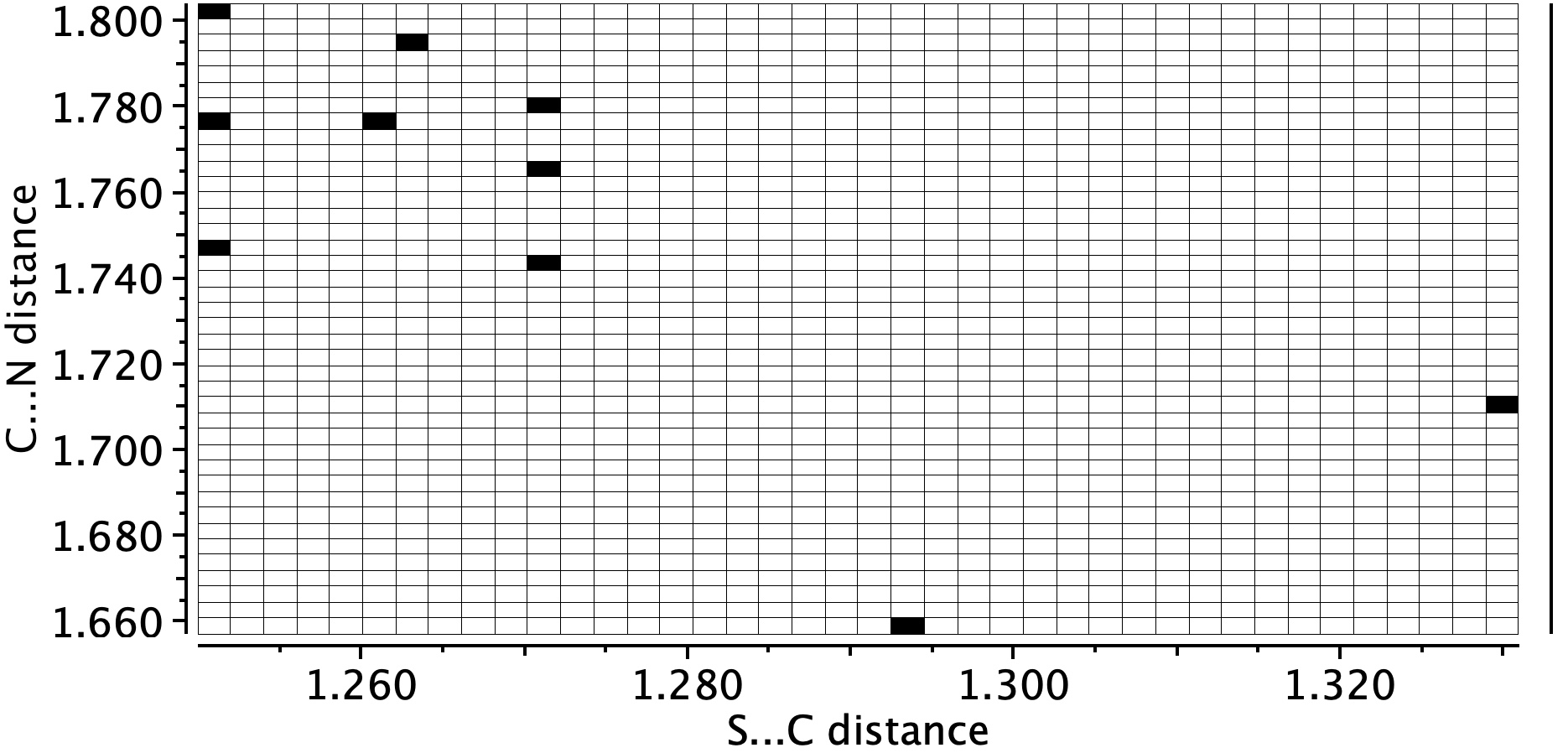
- A pesquisa da estrutura cristalina de unidades com s… c… n em que o n tem apenas um vínculo revela zero hits, mas pesquisas de s… c… nh revelam nove hits, com distâncias s… c na faixa de 1,74-1,80å e c… n distâncias na região 1.25-1.27 & aring. A distância CN relatada é de 1,251 e aring, confirmando que, quando ligado à proteína, o grupo ciano é substituído por um grupo SC = NH e, portanto, é claramente um componente importante do modo de ação do paxlovid.
- A conformação de Paxlovid é, em um aspecto Diagrama da Wikipediacomo mostrado abaixo. Isso implica o t-butil grupo (à esquerda) como bem separado do pirrolidinona sistema de anel à direita da molécula.
De fato, os dois grupos são adjacentes, sendo mantidos nessa conformação provavelmente uma combinação de forças de dispersão fracas e uma contribuição da proteína circundante na estrutura cristalina. Isso é mostrado mais graficamente pelo diagrama NCI (interração não covalente) abaixo (doi: 10.14469/hpc/9964), onde as áreas verdes da região entre os dois grupos (aneladas em vermelho) representam interações estabilizadoras entre eles. Você também pode identificar outras regiões verdes/ciano, indicando ligações fracas de hidrogênio fracas entre os grupos CH e o oxigênio!
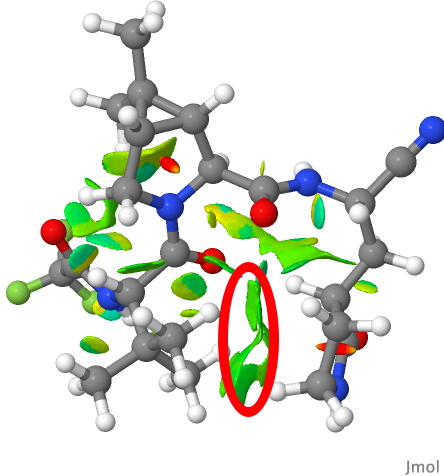
Análise PAXLOVID NCI
Há apenas um pequeno número de estruturas cristalinas de pequenas moléculas contendo o motivo SC = NH. Vou tentar descobrir como isso é comum nas estruturas proteicas-ligantes.
‡ Existem muitas ferramentas para executar esta operação. Eu usei o seguinte procedimento. Eu baixei o arquivo PDB (https://information.rcsb.org/obtain/7vh8.cif), abriu no CSD Mercury, selecionou o ligante (identificando o CF3 grupo e clicando em um átomo), inverteu a seleção para que tudo, menos o ligante, fosse selecionado e usando editar/estrutura, excluí os átomos selecionados, deixando apenas o ligante.
POSTRIPT
The cyanopyrrolidine group comparable to in Paxlovid is nicely often called a selected probe.(cite)10.1039/D1MD00218J(/cite),(cite)10.1021/jacs.0c04527(/cite),(cite)10.1021/acschembio.0c00031(/cite) CovalentInDB is a complete database Facilitar a descoberta de tais inibidores covalentes (citação) 10.1093/nar/gkaa876 (/cite) e está disponível aqui. Há também um programa chamado DataWarrior Isso é potencialmente capaz de encontrar essas sondas.
Relacionado
Esta entrada foi publicada no sábado, 13 de novembro de 2021 às 16:41 e é arquivada em QuímicoAssim, cristal_structure_miningAssim, Em geralAssim, Química interessante. Você pode seguir qualquer resistência a esta entrada através do RSS 2.0 alimentar. Você pode Deixe uma respostaou trackback do seu próprio website.






