A clonagem de DNA é onde começa a inovação biotecnica. De enzimas produzidas sinteticamente a novas variedades de culturas, cepas microbianas, materiais biodegradáveis, biocombustíveis e muito mais – tudo começa com técnicas de clonagem de DNA.
O planejamento de um experimento de clonagem de DNA pode ser esmagador sem entender as muitas estratégias disponíveis para otimizar os fluxos de trabalho. Este artigo compara diferentes abordagens aos métodos de clonagem molecular e de montagem de DNA sintéticos, para que você possa fazer escolhas informadas sobre seus experimentos de clonagem e executá -los bem.
O que se entende por clonagem de DNA?
A clonagem de DNA (também chamada de clonagem molecular) é um termo guarda -chuva para técnicas de laboratório usadas para criar uma população de organismos que carregam a mesma sequência de DNA (clones). (1)
Na sua essência, a clonagem de DNA envolve três aspectos principais:
- Inserir uma sequência de DNA de interesse em um vetor;
- Transformar o vetor no sistema host;
- Utilizando os sistemas de expressão do host para produzir a proteína alvo.
O fluxo de trabalho geral inclui as seguintes etapas descritas na Figura 1.
Figura 1. Fluxo de trabalho básico para técnicas de clonagem de DNA. (2)
Escolhendo seu vetor
Os vetores são moléculas circulares de DNA que carregam sua sequência de interesse de DNA. Existem dois tipos funcionais principais:
- Vetores de clonagem: Esses vetores normalmente contêm elementos essenciais, como uma origem da replicação, vários locais de clonagem para inserção de DNA e um gene de resistência a antibióticos para selecionar as células que transportam o vetor.
- Vetor de expressão: Esses vetores contêm elementos reguladores específicos do hospedeiro, como sequências de promotor e terminador que acionam a expressão da proteína alvo.
Plasmídeos bacterianos são os vetores mais comuns. Outros tipos incluem vetores virais (bacteriófagos), cromossomos artificiais bacterianos (BACs), cromossomos artificiais de levedura (YACs) e vetores específicos de células de mamíferos. (3)
Projetando sua sequência de interesse
Sua sequência de interesse de DNA pode ser sintetizada artificialmente ou isolada usando a reação em cadeia da polimerase (PCR).
Você pode usar um único fragmento de DNA ou montar vários fragmentos para o seu experimento de clonagem. Se você optar por montar vários fragmentos, é mais conveniente e mais rápido fazer isso sinteticamente. Essa abordagem elimina a necessidade de ampliar ou reunir regiões mais curtas de vários modelos, simplificando o processo significativamente.
Além disso, você também pode adaptar sua sequência para ter recursos como locais de enzima de restrição, grupos de fosfato ou sobreposições de sequência complementares para facilitar o processo de montagem. Essas considerações de design mudarão de acordo com a forma como você pretende criar sua construção de DNA-vetor (também conhecida como vetor recombinante).
Criando seu construto de vetor de DNA
Criar um construto de vetor de DNA, ou vetor recombinante, envolve a combinação da sua sequência de interesse de DNA (sua inserção) com o vetor escolhido. Uma abordagem comum é o método ‘corte e pasta’, que utiliza enzimas de restrição e ligase de DNA para se juntar ao DNA e ao vetor.
Técnicas mais recentes, como clonagem sem costura e montagem de portão douradas, ignoram a necessidade de enzimas de restrição, mas requerem design avançado dos fragmentos de DNA. Compreender os pontos fortes e as limitações de cada método é essencial para selecionar a melhor abordagem para sua pesquisa.
Nas seções a seguir, descreveremos e compararemos esses métodos para ajudá -lo a tomar a decisão mais informada.
Clonagem de ponta-brisa
A clonagem de ponta robusta é um método rápido e direto que usa enzimas de restrição para criar pontas robustas nos fios de DNA, que não têm bases salientes. No entanto, o método não é direcional, o que significa que o DNA pode ser inserido em orientações aleatórias. Isso o torna menos adequado para montar vários fragmentos, pois um ou mais dos fragmentos podem ser inseridos na direção errada.
Para melhorar a eficiência da clonagem de ponta robusta, é melhor preparar o DNA com extremidades de 5′-fosfato, pois esses grupos são essenciais para a ligação com o vetor. Além disso, a remoção de 5′-fosfatos do vetor impede que ele se auto-lige, garantindo que ele se junte apenas à inserção.
TA clonagem
A clonagem de TA é outro método rápido e fácil. Em vez de enzimas de restrição, a polimerase TAQ adiciona saliências de nucleotídeos A e T, criando extremidades pegajosas nos fios de DNA e vetor. Este método não é direcional, tornando-o mais adequado para construções de fragmentação única.
Os fragmentos de DNA sintético podem ser ordenados como IS, e A-Overhangs pode ser adicionado posteriormente usando a polimerase TAQ. Para fragmentos amplificados por PCR, A-overhangs pode ser incluído diretamente em seus primers.
Clonagem da enzima de restrição
Da mesma forma que a clonagem de TA, a clonagem da enzima de restrição cria saliências de nucleotídeos curtas nos fios de DNA e no vetor. Embora esse método ofereça clonagem direcional, ele não é best para montar vários fragmentos, pois se baseia em locais específicos de reconhecimento de enzimas para cortar e unir o DNA.
Para aprimorar esse método, projete fragmentos de DNA com locais de reconhecimento para enzimas de restrição e adicione 4-5 bases extras para melhorar a ligação à enzima.
Golden Gate
O método Golden Gate permite a montagem de construções contendo vários fragmentos de DNA sem deixar “cicatrizes” (bases residuais que podem afetar a expressão de proteínas). Este método é particularmente adequado para montar fragmentos com complexidades estruturais internas.
No entanto, os locais de reconhecimento de enzimas devem ser cuidadosamente colocados para evitar cortes indesejados, e sequências de saliências semelhantes podem reduzir a eficiência, causando ligação não intencional.
Clonagem sem costura
Assim como a clonagem do portão dourado, o método sem costura monta vários fragmentos de DNA sem deixar cicatrizes. No entanto, é sensível a complexidades de sequência, como regiões ricas em GC, sequências repetitivas ou estruturas secundárias, que podem prejudicar o processo de montagem.
Clonagem de gateway
O método de clonagem de gateway (Thermo Fisher Scientific) facilita a transferência de fragmentos de DNA para vetores de destino usando locais homólogos (ATT). Esse processo é rápido e eficiente, e não requer ligase.
Um resumo de cada método pode ser visto na Figura 2, e os prós e contras de cada método são descritos na Tabela 1.
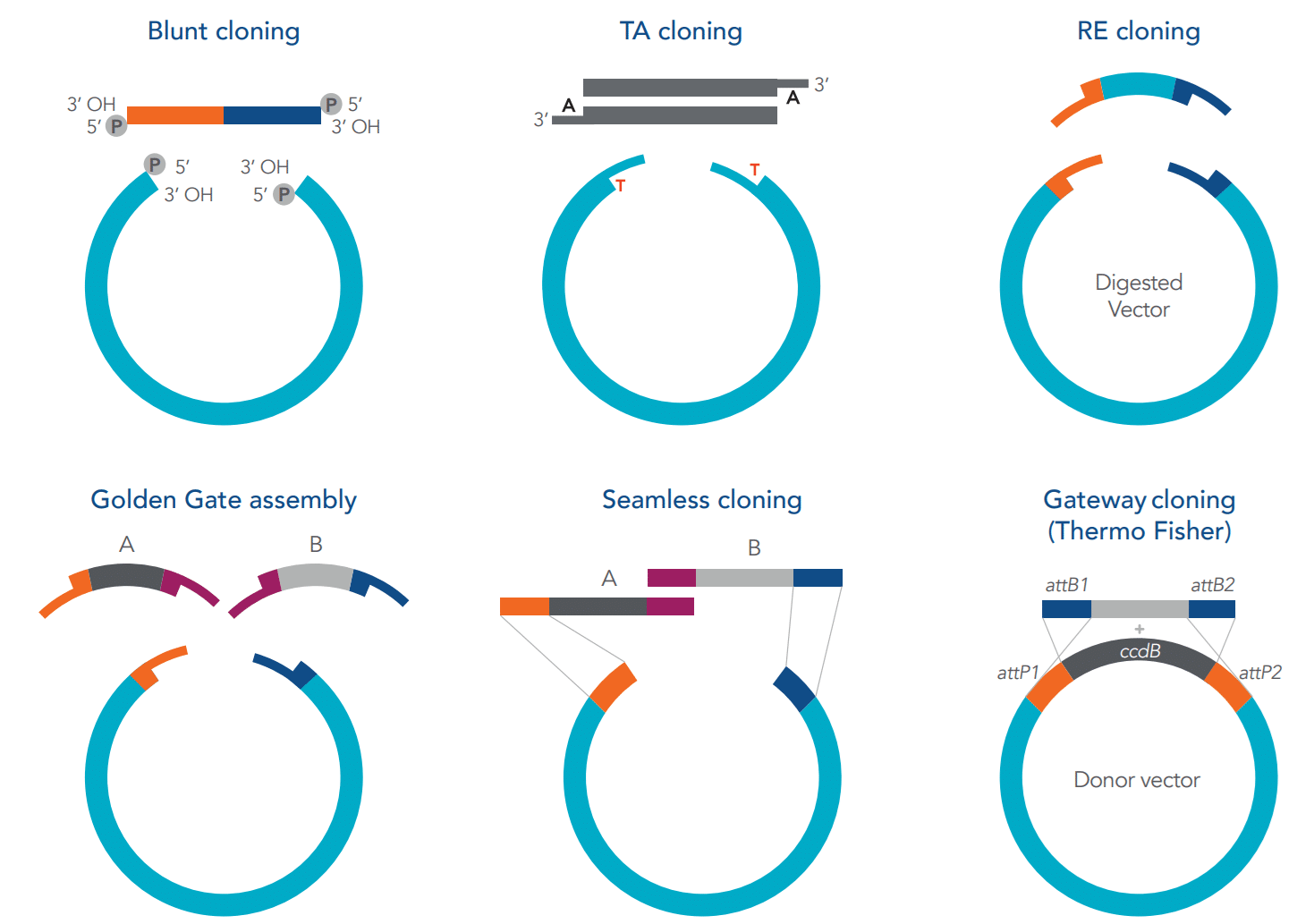

Figura 2. Resumo de cada método de clonagem. (2)
Tabela 1. Resumo dos métodos de clonagem. (2)
|
Clonagem de ponta-brisa TA clonagem |
Rápido e fácil Sem etapas extensas de preparação ou design |
Clonagem não direcional Muitas vezes requer mais triagem Não é adequado para montar construções maiores |
|
Clonagem de enzima de restrição tipo II (pontas pegajosas) |
Clonagem direcional com alta eficiência Locais de corte de enzima de restrição exclusivos garantem a ligação correta Enzimas e buffers de restrição amplamente disponíveis |
Requer locais de corte de enzima de restrição em inserção e vetor Folhas “cicatrizes” ou bases residuais no construto |
|
Clonagem de enzima de restrição do tipo IIS (montagem de portão dourado) |
Clonagem sem cicatrizes como enzimas do tipo IIS se separam do web site de reconhecimento Pontas pegajosas para clonagem e ligação altamente eficientes Bom método para montar vários fragmentos |
Requer locais de corte de enzima de restrição em inserção e vetor Pode resultar em ligação não específica se as saliências de fragmentos forem semelhantes (diferir apenas por uma base ou duas) |
|
Clonagem rápida de fragmentos únicos ou múltiplos em uma única reação de montagem Não requer websites de restrição Altamente eficiente |
Requer sobreposições de 15 a 40 pares de bases entre sequências de fragmentos, inserções e vetores |
|
|
Troca rápida de inserções entre vetores Grande seleção de plasmídeos compatíveis com gateway |
Requer websites att em pontas de flanqueamento de fragmentos, então deixa “cicatrizes” Não pode ser usado para montar vários fragmentos |
Projetando e montando fragmentos de DNA sintético
Por que montar vários fragmentos de DNA?
A montagem de múltiplos fragmentos permite que você atinja comprimentos de sequência de até 3 kb. Essas seqüências mais longas podem acomodar uma variedade de elementos, incluindo regiões codificadoras de proteínas, elementos reguladores, partes biológicas, fragmentos de anticorpos, regiões genômicas e muito mais.
Os desafios da montagem de fragmentos de DNA
Grandes construções geralmente vêm com complexidades de sequência que podem interferir na síntese, montagem ou uso remaining de fragmentos de DNA. As complexidades comuns e suas estratégias de gerenciamento estão listadas abaixo e expandidas na Tabela 2.
- Extremos no conteúdo de GC: gerencie com uma sequência estabilizadora.
- Repita regiões: gerencie criando sub-fragmentos.
- Estruturas secundárias: gerencie com otimização de códon.
Tabela 2. Resumo das estratégias para reduzir as complexidades das sequências de DNA. (2)
|
Adicionando sequências estabilizadoras |
Dividindo-se em sub-fragmentos |
|
|
Adicione bases extras ao terminal de fragmentos |
Crie sub-fragmentos da sequência unique |
Modificar sequências de DNA enquanto preservam a sequência de aminoácidos |
|
Mais eficaz quando as complexidades estão em extremidades terminais |
Mais eficaz quando as complexidades internas são espaçadas e podem ser separadas |
Mais eficaz para complexidades espalhadas por toda a sequência de DNA |
|
Bases estabilizadoras podem precisar ser removidas |
Sub-fragmentos precisam ser remontados |
A sequência de DNA otimizada será diferente da sequência unique |
Considere usar ferramentas on -line para ajudar a projetar experimentos de clonagem
O software program de manipulação de sequência de DNA simplifica bastante o design de experimentos de clonagem. Essas ferramentas podem anotar sequências, gerar mapas vetoriais, analisar conteúdo de GC, identificar motivos estruturais, calcular temperaturas de fusão, localizar locais de enzima de restrição e visualizar construções clonadas. Isso ajuda a garantir que você não compre uma sequência que não seja viável.
O SCITOOLSTM da IDT oferece um modo de triagem de complexidade que verifica seqüências de entrada para os três tipos de complexidades descritas acima. Ele destaca regiões problemáticas dentro de uma sequência de DNA e fornece sugestões de melhoria.
Exemplo: Reduza a complexidade adicionando uma sequência estabilizadora
Aqui está um exemplo de como as sequências estabilizadoras podem ajudar a reduzir as pontuações da complexidade. A sequência de teste continha uma região com alto teor de GC na extremidade 3 ‘da sequência. A ferramenta de complexidade identificou esse problema e sinalizou a sequência como não sendo viável. Uma vez adicionado a sequência estabilizadora (em vermelho), a pontuação da complexidade foi reduzida a moderada, tornando a sequência viável para compra (Figura 3).
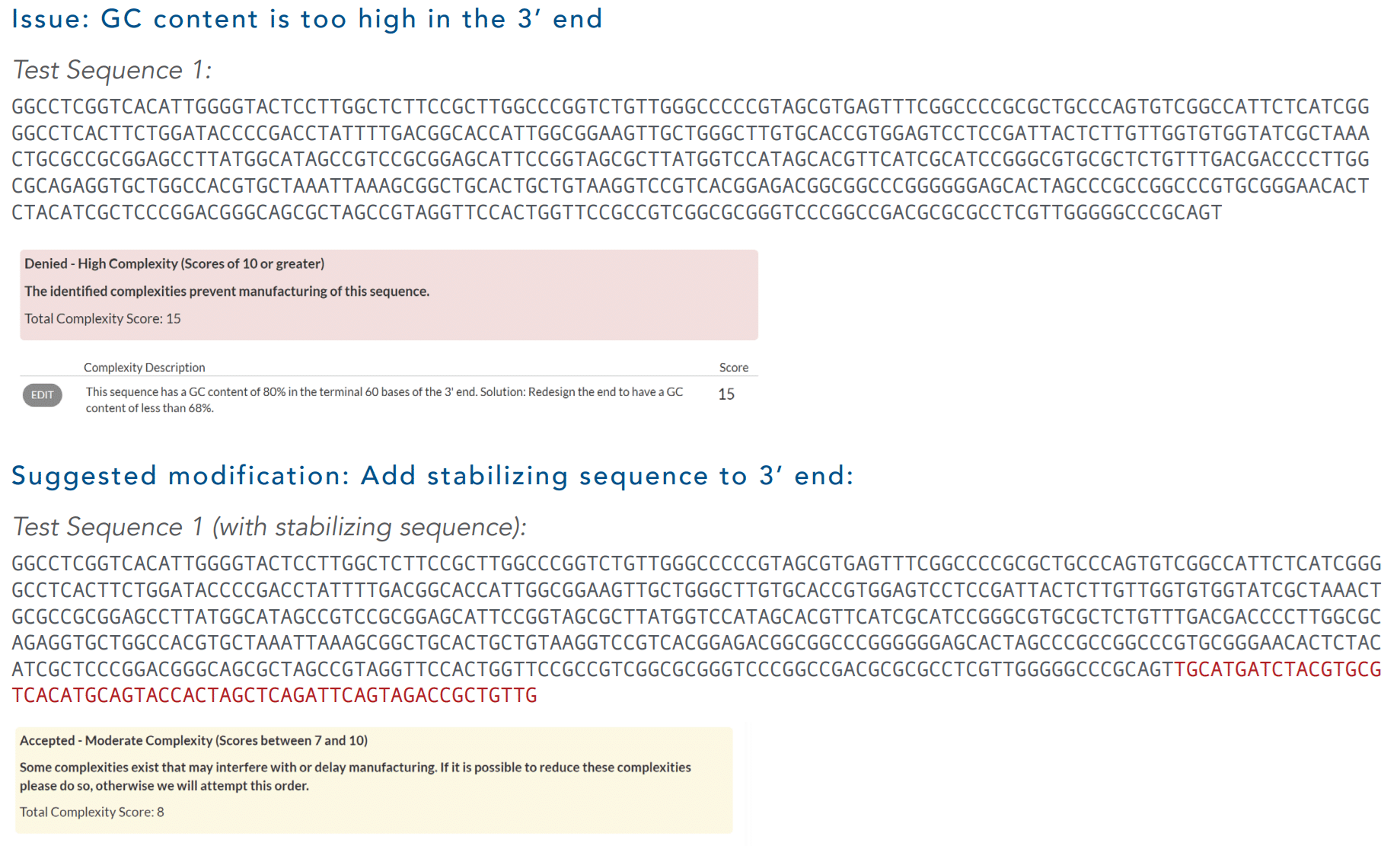

Figura 3. Exemplo de redução da pontuação da complexidade de um Fragmento do gene Gblocks (IDT) Adicionando uma sequência estabilizadora.
A clonagem de DNA resumida
A clonagem de DNA é a pedra angular da biotecnologia, oferecendo diversos métodos para atender a várias necessidades de pesquisa. Com tantas opções disponíveis, entender esses métodos é essencial para superar os desafios e alcançar resultados confiáveis.
Para informações mais detalhadas sobre as técnicas de clonagem, design de sequência, solução de problemas e ferramentas inovadoras da IDT, discover o Guide de clonagem de DNA IDT. É o seu recurso preferido para tornar a clonagem de DNA mais eficiente e eficaz.
Inscreva -se Idt Insider Para ser um dos primeiros a aprender sobre os anúncios de novos produtos, registre -se para os próximos seminários on -line e até ler os holofotes convincentes dos funcionários.
Referências
- Bertero A, Brown S e Vallier L. (2017). Capítulo 2, Métodos de Clonagem. Em Métodos de ciências básicas para pesquisadores clínicos (pp. 19-39). Educational Press
- Guide de clonagem de DNA. (2022). Tecnologias de DNA integradas
- Clark DR, Pazdernik NJ, McGehee MR. (2019). Capítulo 7, Genes de clonagem para biologia sintética. Em Biologia Molecular (pp. 199-239). 3ª ed







