No last da década de 1980, como relembrei aqui(cite)10.59350/g4j62-4xk50(/cite) o equipamento necessário para visualização molecular em tempo actual como ficou conhecido ainda period caro, exigindo sistemas customizados como estações de trabalho Evans e Sutherland PS390. Um grande avanço na disponibilização geral de tais técnicas em equipamentos menos especializados foi alcançado por Roger Sayle(cite)10.1016/s0968-0004(00)89080-5(/cite), então trabalhando no Imperial School por volta de 1990 e usando uma estação de trabalho Silicon Graphics . Ele otimizou bastante os algoritmos de renderização criando um programa chamado RasMol (depois de suas iniciais), o que significava que tais visualizações também poderiam ser alcançadas muito rapidamente, mesmo em um computador pessoal. A mudança da tecnologia de exibição vetorial (o PS390) para gráficos raster/bitmap permitiu representações de preenchimento de espaço de moléculas contendo centenas, se não milhares de átomos – e, por sua vez, permitiu que a nova World Broad Net explorasse a técnica. /citar)
Embora Rasmol seja muito ainda por aítambém proporcionou inspiração para programas sucessores, como Jmol (baseado em Java) e JSMol (baseado na linguagem Javascript incorporada em todos os navegadores modernos). Existem agora muitos artigos na literatura descrevendo este programa. Em 2008, o primeiro publish neste weblog descreveu como executá-lo em uma instância do WordPress(cite)10.59350/pq7ds-gqr71(/cite).
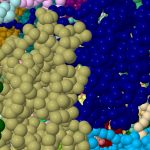
Agora foi alcançado um novo marco na visualização molecular – a capacidade de exibir 3 milhões de átomos! Bob Hanson acaba de lançar Jmol/JSmol 16.1.51 que suporta o formato de arquivo BinaryCIF. Um exemplo do poder do programa e deste novo formato é ilustrado com a proteína 8glv(cite)10.2210/pdb8GLV/pdb(/cite) que contém 3 milhões de átomos (o próprio arquivo bcif tem apenas 47,4 Mb).
O script Jmol/JSmol para carregá-lo é:
t = now();
set autobond false;
load =8glv.bcif filter "*.CA";
spacefill on;
shade chain;
print now
e a renderização actual leva apenas 10 a 20 segundos. Você pode ver nas imagens abaixo‡ que quando ampliado, ele realmente mostra átomos individuais! Quem sabe qual é o limite prático de átomos, mas é quase certo que é superior a três milhões! E pode até ser possível num telemóvel!
‡OK, você está perguntando por que não carreguei 8glv nesta página? Bem, primeiro preciso atualizar o JSmol neste website e encontrei um problema que precisa ser corrigido.
Pós-escrito. Estamos chegando lá. Abaixo está uma captura de tela desta proteína usando um iPhone 15 e qualquer um dos navegadores Safari, FireFox, Chrome ou Edge by way of este hyperlink (cerca de 12 segundos no iPhone, ~30 segundos em um iMac 2019, ~12 segundos em um M1 Mac Studio). Ele ainda gira razoavelmente bem!