Moléculas do ano -2022. Problemas de dados!
A lista de moléculas do ano já está disponível em Notícias C&E (mas é necessário ter uma conta para visualizar a lista, ao contrário dos anos anteriores).♣ Esses três chamaram minha atenção:
- Elétron em um cubo: Síntese e caracterização do perfluorocubano como aceptor de elétrons,(cite)10.1126/science.abq0516(/cite). eu já escrito sobre este sistema e não discutirei mais o assunto, exceto para observar que este ficou no topo da enquete!
- Síntese de modelo Vernier de nós moleculares(citar)10.1126/science.abm9247(/cite)
- Megalo-Cavitands: Síntese de Acridane(4)arenos e Formação de Cavitands Grandes e Profundos para Captação Seletiva de C70(citar)10.1002/anie.202209885(/citar)
Os dois últimos são exemplos de grandes moléculas tridimensionais com propriedades incomuns. O segundo é um exemplo de trevo de trevos, chamado nó de tríscele e eu estava muito interessado em obter suas coordenadas para poder inspecionar o nó. Pensei em resumir aqui os procedimentos hierárquicos que podemos tentar para adquirir tais dados.
- O método mais moderno de aquisição de dados associados a um artigo é inspecionar a lista de citações no remaining do artigo. A tendência encorajada pelos princípios de dados FAIR sugere que se tais dados tiverem um DOI associado (como de facto o próprio artigo tem), então este DOI deve ser citado nas citações tal como os próprios artigos. Este conceito também é conhecido como tratar dados como um cidadão de primeira classe dos processos acadêmicos. Neste caso, nenhum dado foi associado às 81 citações listadas em 10.1126/science.abm9247
- O método predominante desde 1996 tem sido o próximo obtain de qualquer ESI. Isso está ligado aqui. Não posso deixar de observar que o formato PDF não é otimizado para dados, mas é melhor que nada. Este PDF tem 114 páginas, e eventualmente encontra-se o seguinte na p 103: estruturas e energias correspondentes carregadas no banco de dados do Github (https://github.com/kjhstenlid/AshbridgeVernier2022/). O Github é conhecido como repositório de software program, mas seu uso como repositório de dados é incomum. Portanto, nenhum DOI recebe esses dados (o que explicaria por que não está listado nas citações do artigo). Aqui se aprende com o leia-me que ele contém Estruturas de nós moleculares em formato de arquivo cif para os nós Verner e Sheild.
- Para obter esses dados é preciso fingir que é um código e baixar o arquivo do CEP. O arquivo CIF encontrado lá, entretanto, apresenta um erro deadly ao tentar carregar em um visualizador CIF como o Mercury: A leitura da célula do Cif falhou, não foi possível recuperar ‘_cell_length+a’. O CIF claramente não é gerado a partir de um programa de análise cristalográfica, mas sim de um programa de modelagem e é claramente inválido como CIF.
- Agora é preciso voltar atrás para ver se o arquivo CIF pode ser resgatado usando um editor de texto. Isso não é trivial, mas cerca de 10 minutos de edição finalmente produzem um arquivo que pode ser visualizado.
- Aqui está a estrutura 3D (clique na imagem para visualizar).
Agora vamos aos Megalo-Cavitands (ou não). Assim como acima, acaba-se em um Arquivo PDF de 49 páginas procurando coordenadas. Lá são obtidas fotos de modelos computados pelo PM6 começando na página 28, mas, infelizmente, aparentemente nenhuma coordenada associada.†
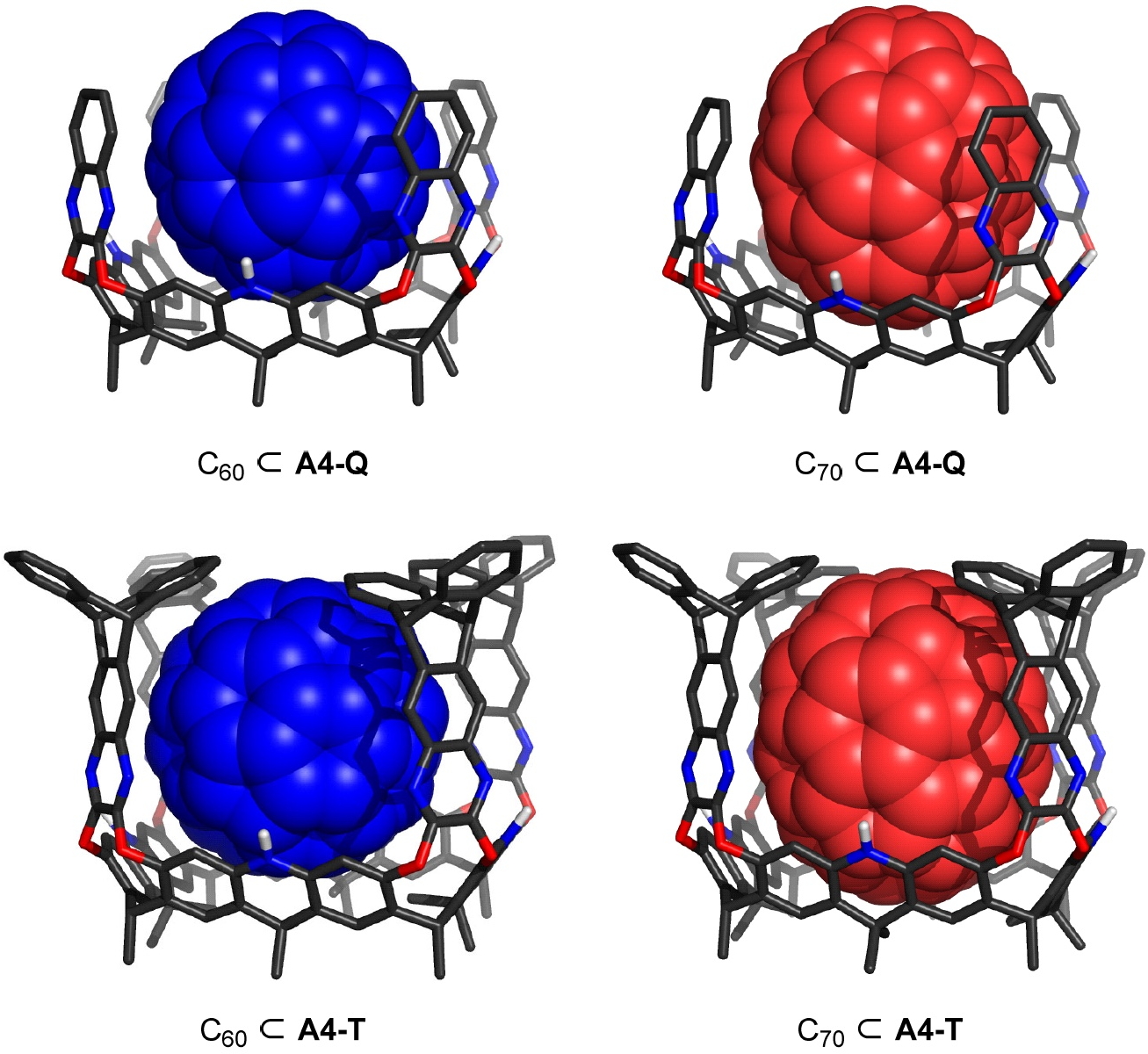
Portanto, não há modelos 3D para mostrar aqui (desculpe, clicar na imagem acima não os produzirá†).
Minha observação remaining deveria ser que quando uma molécula interessante é selecionada para inclusão em por exemplo nas moléculas do ano – 2022, um dos critérios para a sua inclusão é que a disponibilidade de dados completos e FAIR descrevendo as suas propriedades seja um dos critérios essenciais para a seleção.
‡Observo que o método usado para gerar essas coordenadas (PM6) talvez não seja o ultimate; ele não contém termos de atração de dispersão, que provavelmente são importantes na modelagem da complexação hospedeiro-convidado. O método PM7 é muito melhor para esse tipo de coisa! Isto destaca a importância de fornecer dados, neste caso coordenadas 3D. Seria interessante recalcular as dimensões dessas moléculas usando um método que permitisse incluir atrações de dispersão. Para apenas um exemplo, veja aqui.
† Entrei em contato com os autores de (cite)10.1002/anie.202209885(/cite) e descobri que uma referência a um envio de repositório de dados foi omitida do artigo. Os dados estão no DOI: 10.5281/zenodo.6953961 e relatarei separadamente minha análise do efeito da substituição de PM6 por PM7.
♣Veja isto carta aberta sobre mudanças na C&EN.
Esta postagem tem DOI: 10.14469/hpc/12028
Relacionado
Esta entrada foi publicada terça-feira, 13 de dezembro de 2022 às 15h34 e arquivada em Sem categoria. Você pode acompanhar quaisquer respostas a esta entrada através do RSS 2.0 alimentar. Você pode deixe uma respostaou trackback do seu próprio web site.







